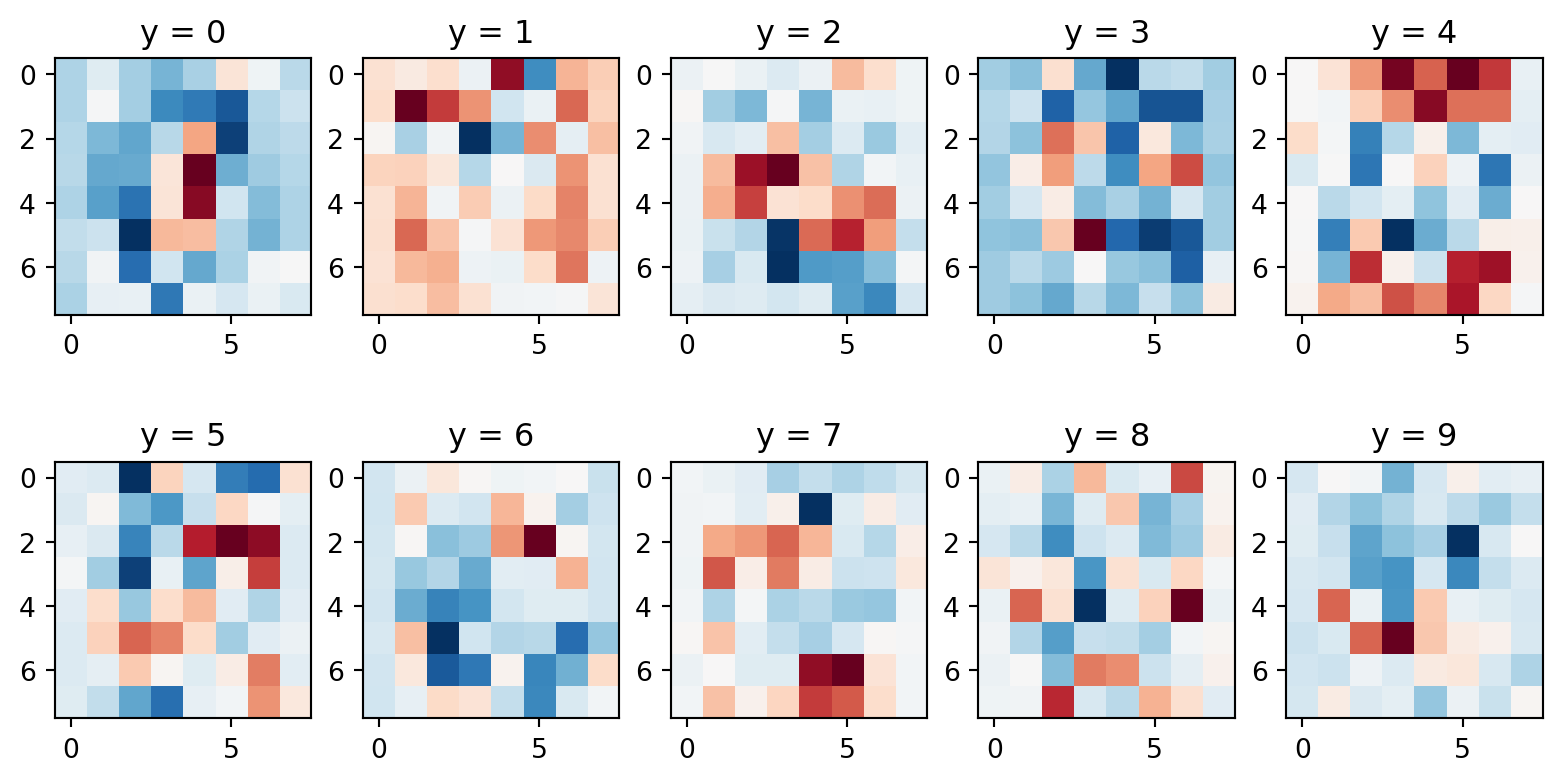
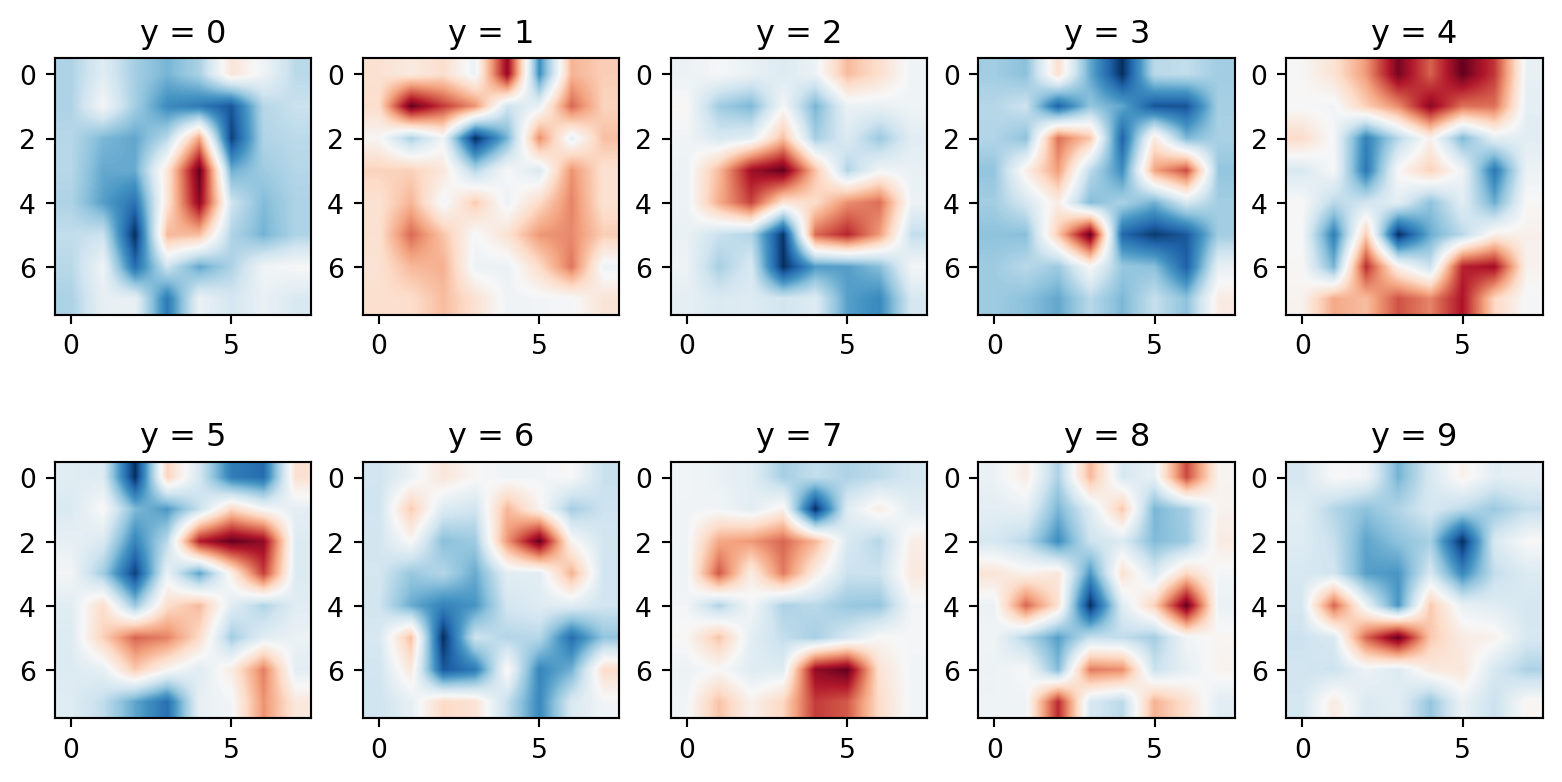
Code
import numpy as np
import matplotlib.pyplot as plt
import seaborn as sns
try:
from palmerpenguins import load_penguins
except ImportError:
! pip install palmerpenguins
from palmerpenguins import load_penguins
# Charger le jeu de données Palmer Penguins
df = load_penguins()
# Ne garder que 'flipper_length_mm' et 'species'
df = df[['flipper_length_mm', 'species']]
# Supprimer les lignes avec des valeurs manquantes (NaNs)
df.dropna(inplace=True)
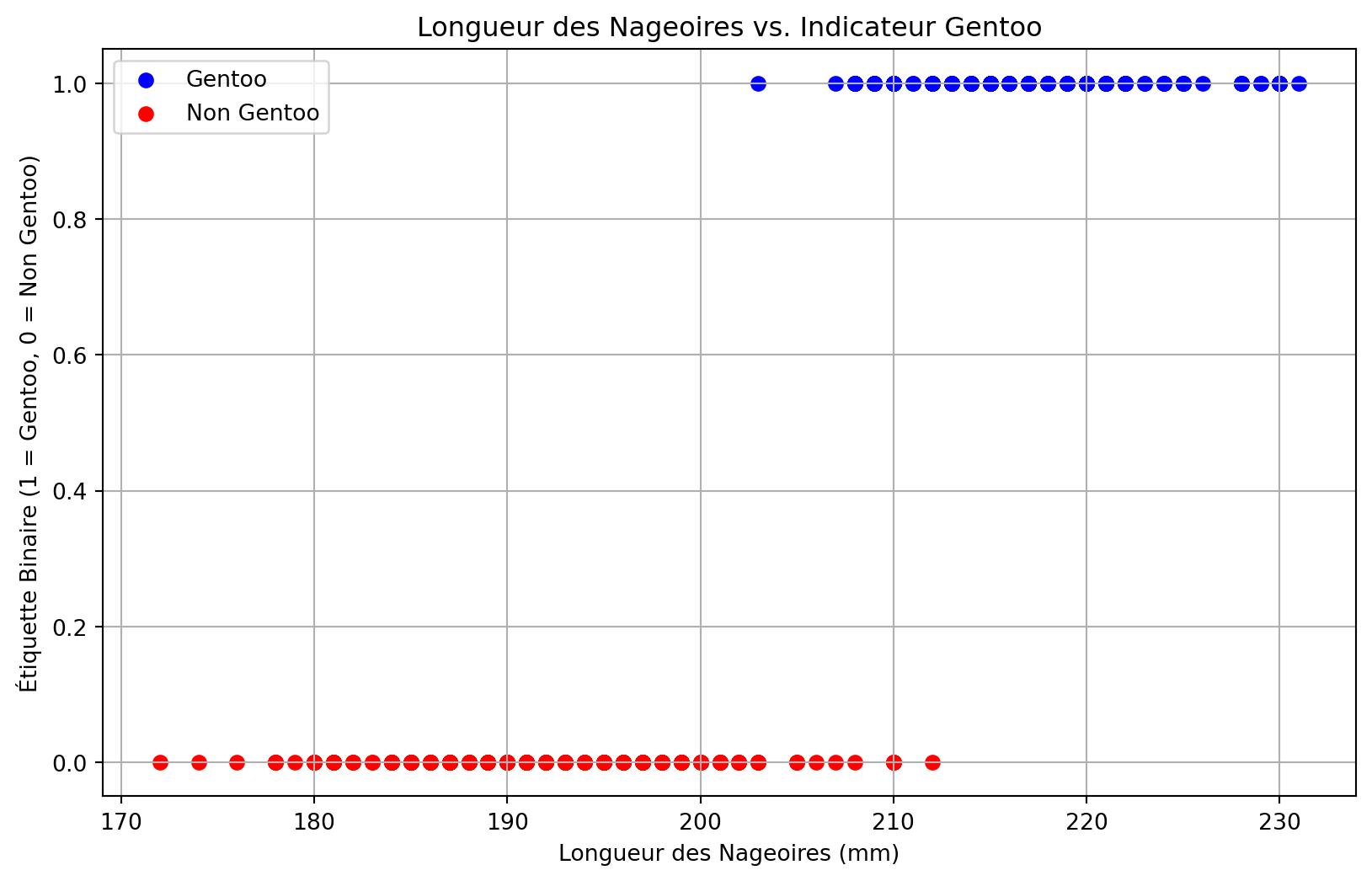
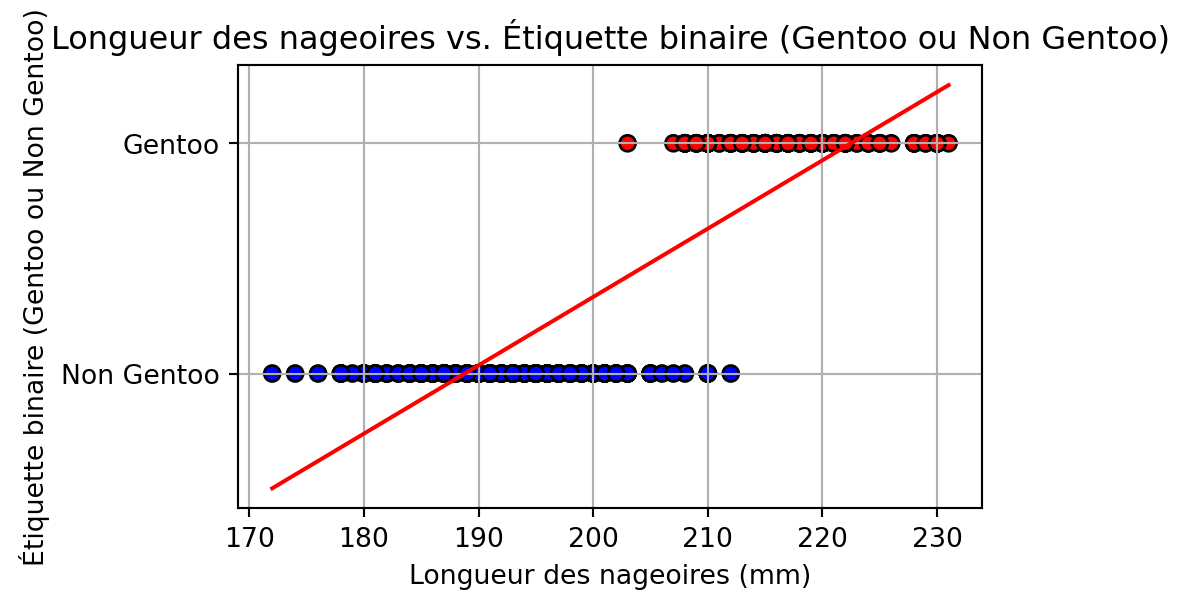
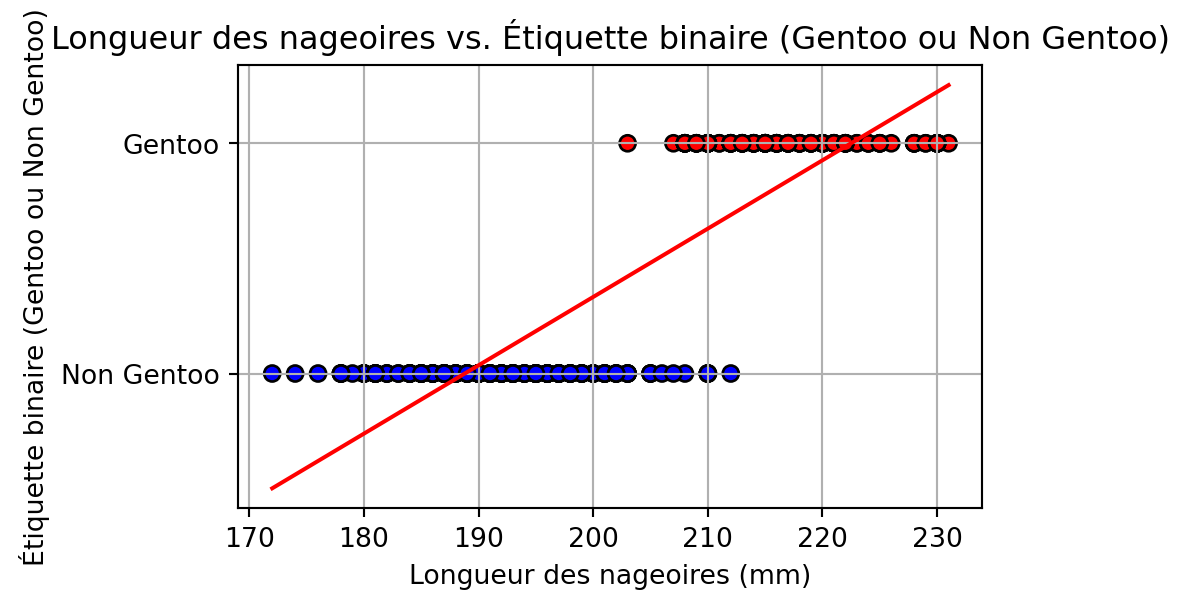
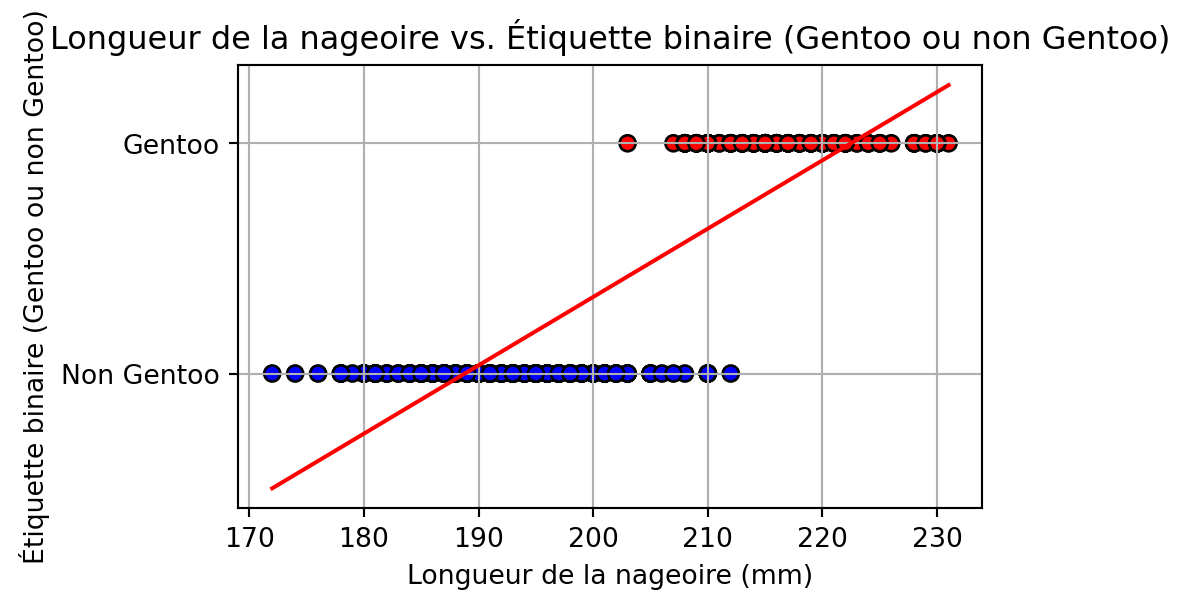
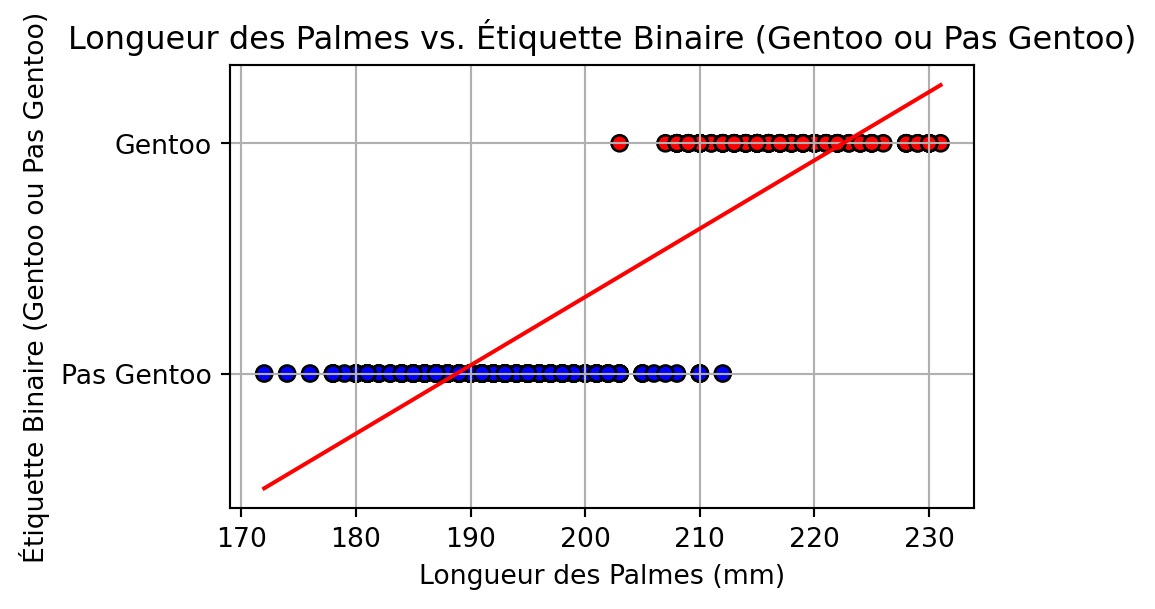
# Créer une étiquette binaire : 1 si Gentoo, 0 sinon
df['is_gentoo'] = (df['species'] == 'Gentoo').astype(int)
# Séparer les attributs (X) et les étiquettes (y)
X = df[['flipper_length_mm']]
y = df['is_gentoo']
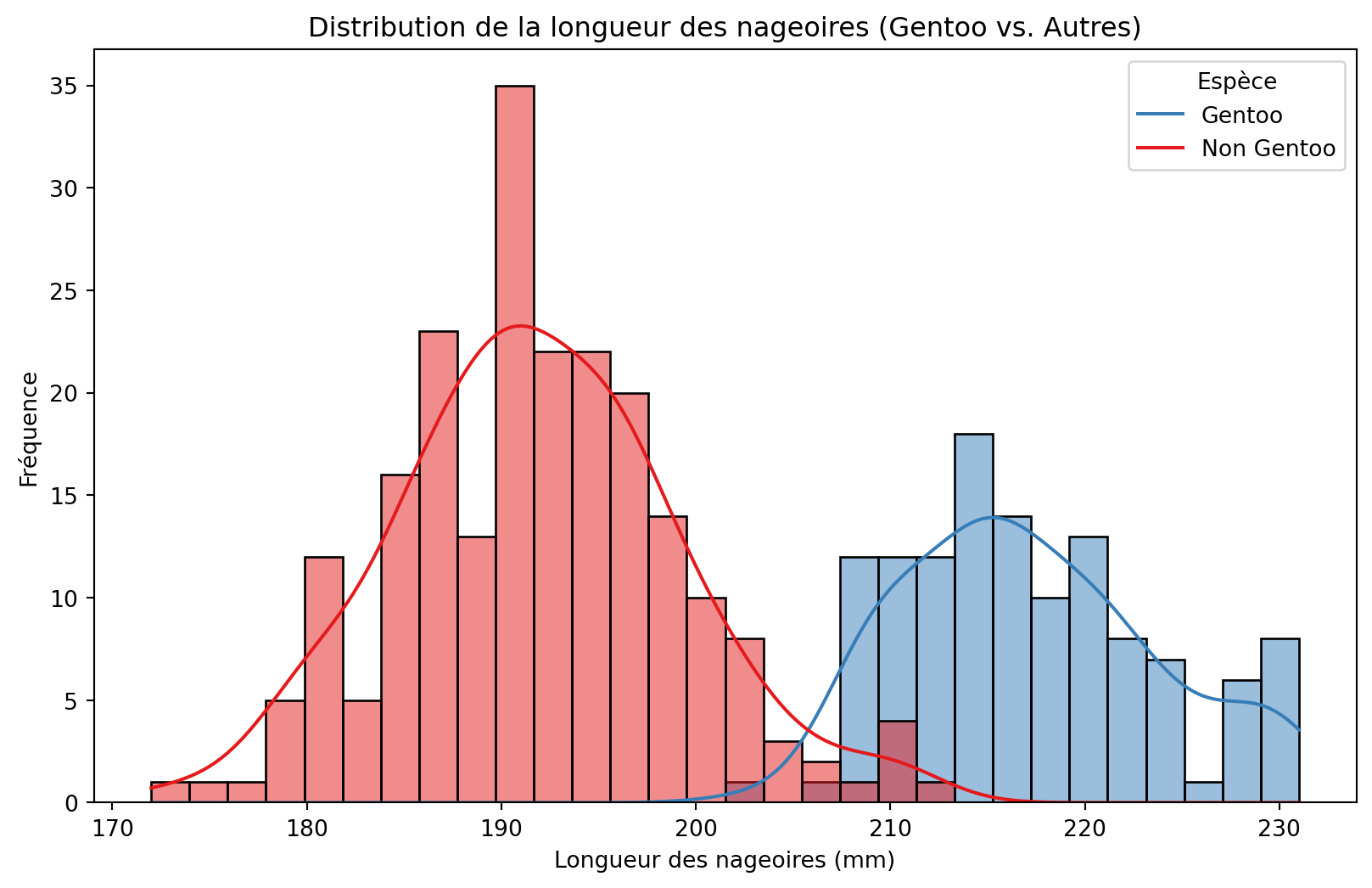
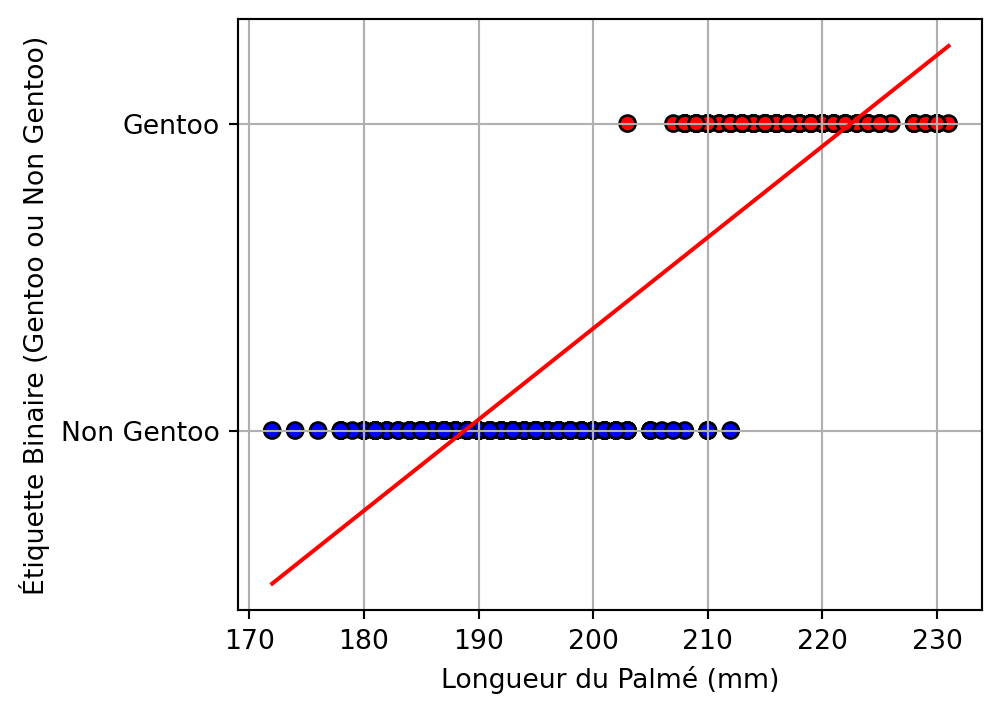
# Tracer la distribution des longueurs de nageoires par étiquette d'espèce binaire
plt.figure(figsize=(10, 6))
sns.histplot(data=df, x='flipper_length_mm', hue='is_gentoo', kde=True, bins=30, palette='Set1')
plt.title('Distribution de la longueur des nageoires (Gentoo vs. Autres)')
plt.xlabel('Longueur des nageoires (mm)')
plt.ylabel('Fréquence')
plt.legend(title='Espèce', labels=['Gentoo', 'Non Gentoo'])
plt.show()